在生命體系中,蛋白質很少單獨發揮其生物學功能,而是依賴於蛋白質-蛋白質相互作用(PPI)來執行復雜的生物學過程。因此,設計能夠特異性靶向和調控 PPI 的蛋白質結合物具有巨大的治療和生物技術潛力。 然而,傳統的蛋白質結合物生成方法,例如免疫接種、抗體庫篩選或定向進化,通常費力費時,並且對靶位的控制有限。
計算蛋白質設計提供了一種強大的替代方案,能夠根據特定靶標和結合位點定製結合物。而早期計算方法(如 Rosetta)雖然嘗試通過物理建模與側鏈優化設計結合界面,但成功率往往不足 0.1%。 進入深度學習時代後,AlphaFold2 等結構預測模型徹底改變了這一局面。它不僅能精準預測單個蛋白的三維摺疊結構,還能模擬蛋白複合物的空間互作方式。
但這仍非真正意義上的「智能設計」。現有方法,如 RFdiffusion 或 ProteinMPNN,依然需要人工設定骨架、對接界面,再用 AlphaFold 來驗證是否合理。 那麼,既然 AlphaFold 已經能理解蛋白結構,那能否讓它自己反向「思考」,直接生成一個與目標完美契合的新蛋白?
基於此,瑞士洛桑聯邦理工學院(EPFL)與麻省理工學院(MIT)的團隊提出了一個用於從頭設計蛋白質結合物的開源自動化流程 BindCraft,其核心思想是通過 AlphaFold2 權重反向傳播幻覺結合劑序列並計算誤差梯度。 研究團隊將 AlphaFold2 的多體版本與神經網絡相結合,通過梯度優化同時生成蛋白的結構、序列與界面。無需高通量篩選、實驗迭代,甚至無需已知結合位點,即可在硅片中生成納摩爾級親和力的從頭結合劑。
實驗結果顯示,BindCraft 在 12 個結構複雜、藥理學意義重大的目標上均取得突破性成果,實驗成功率涵蓋 10%–100%,平均成功率達 46.3%。 這意味着,以往需要數百甚至上千次篩選的設計工作,如今只需一次計算即可獲得可用的結合體。
「BindCraft:蛋白質粘合劑設計」現已上線至 OpenBayes 公共教程板塊, 點擊下方鏈接即可一鍵部署。
教程地址:https://go.openbayes.com/FfIMc
Demo 運行
01 Demo 運行階段
1.登錄 http://OpenBayes.com,在「公共教程」頁面,選擇「BindCraft:蛋白質粘合劑設計」教程。
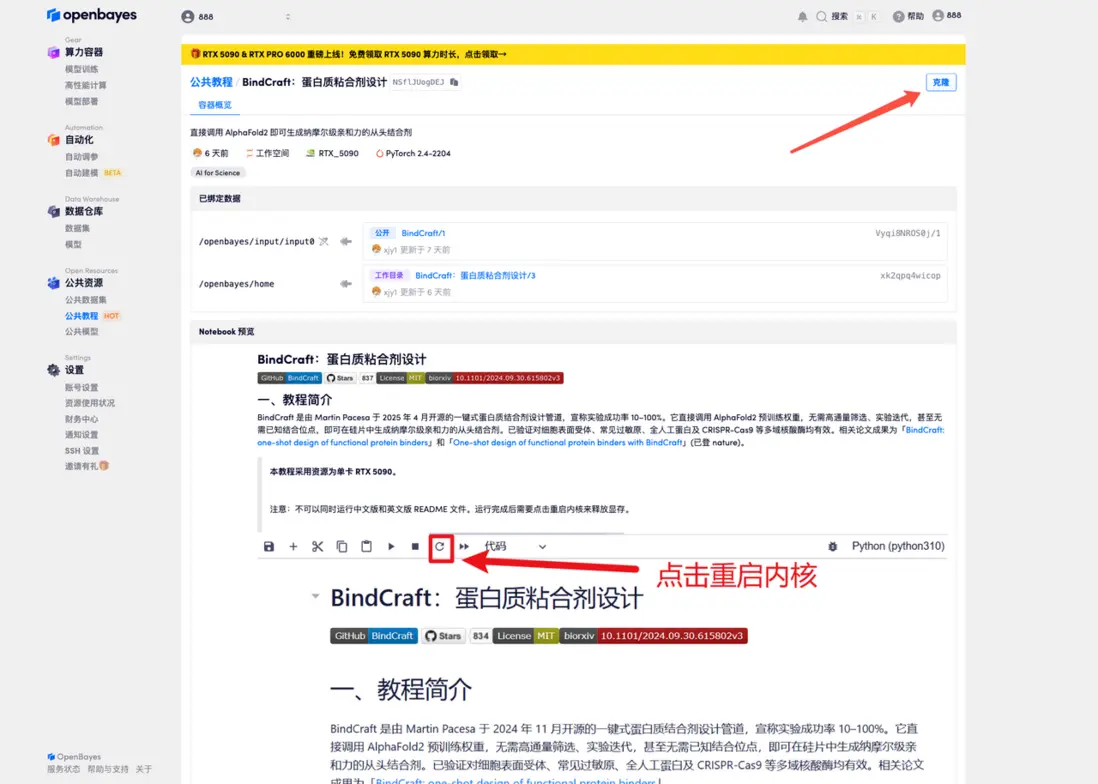
2.頁面跳轉後,點擊右上角「克隆」,將該教程克隆至自己的容器中。
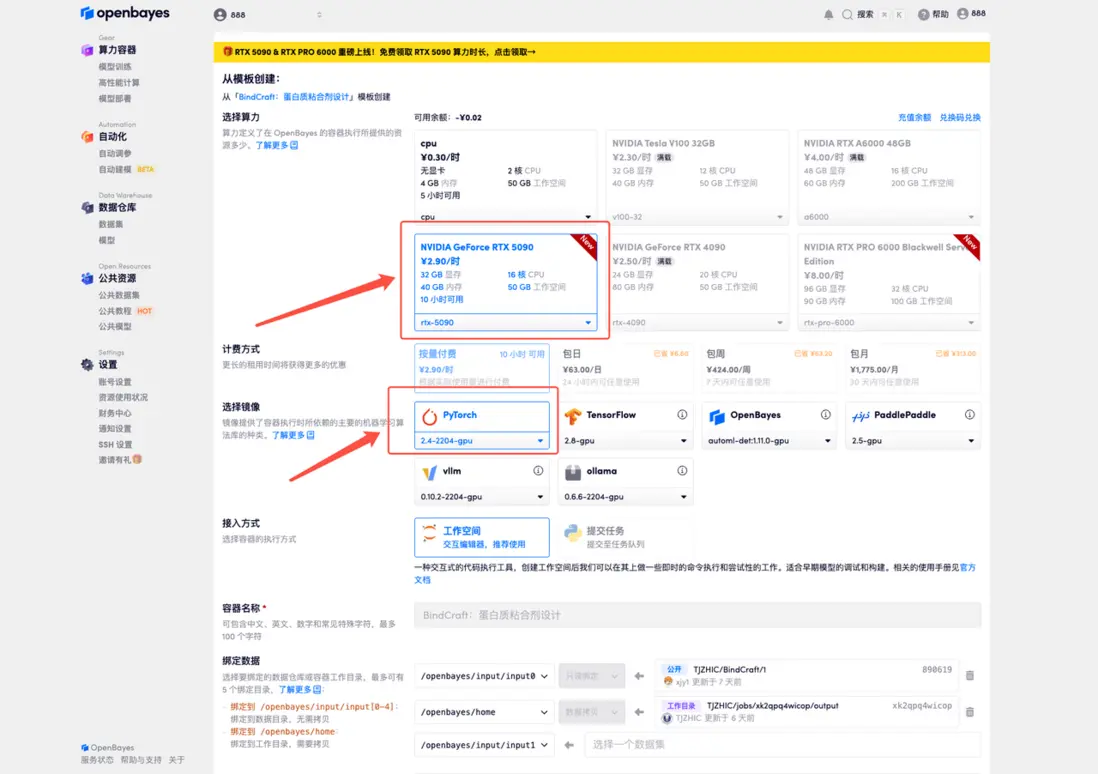
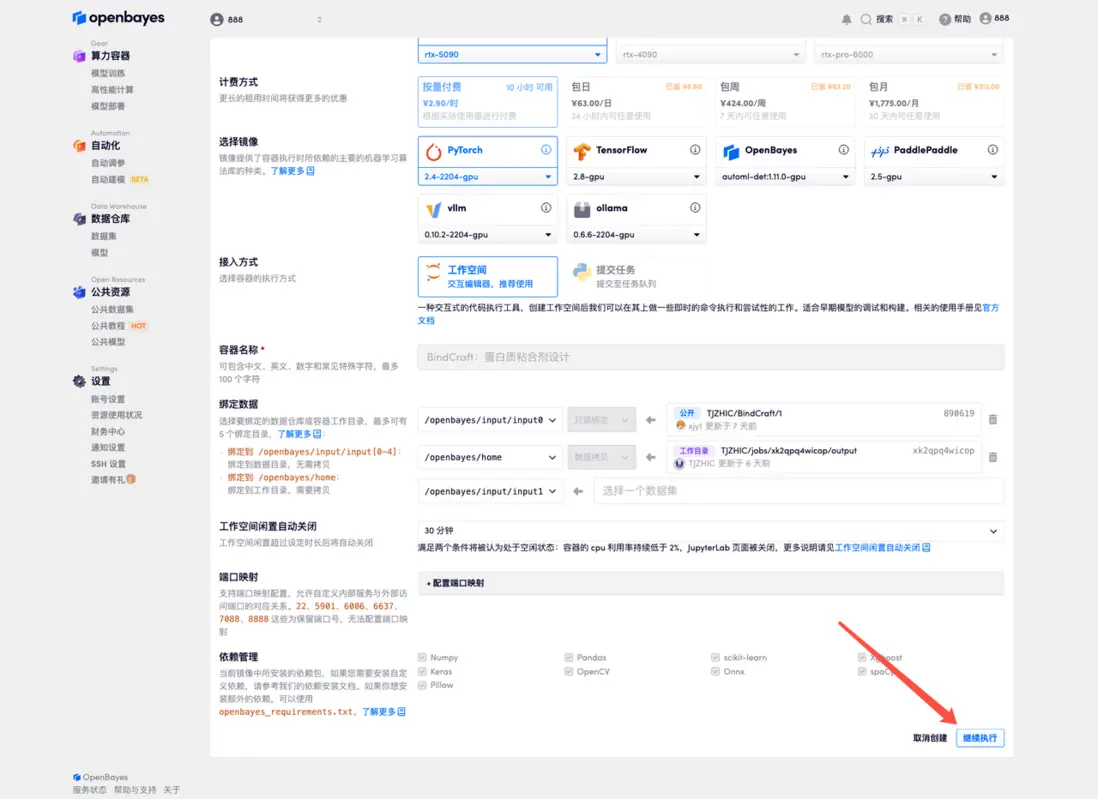
3.選擇「NVIDIA GeForce RTX 5090」以及「PyTorch」鏡像,按照需求選擇「按量付費」或「包日/周/月」,點擊「繼續執行」。新用户使用下方邀請鏈接註冊,可獲得 4 小時 RTX 5090 + 5 小時 CPU 的免費時長!
小貝總專屬邀請鏈接(直接複製到瀏覽器打開):
https://go.openbayes.com/9S6Dr
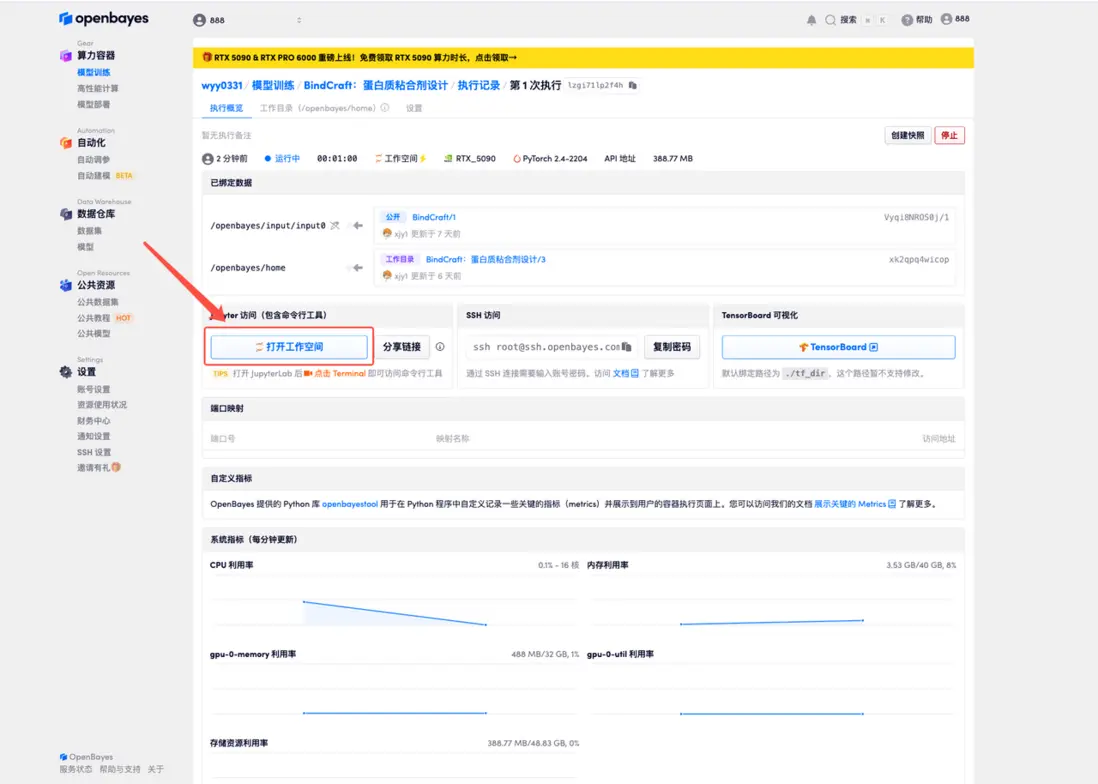
4.等待分配資源,首次克隆需等待 2 分鐘左右的時間。當狀態變為「運行中」後,點擊「打開工作空間」旁邊的跳轉箭頭,即可跳轉至 Demo 頁面。
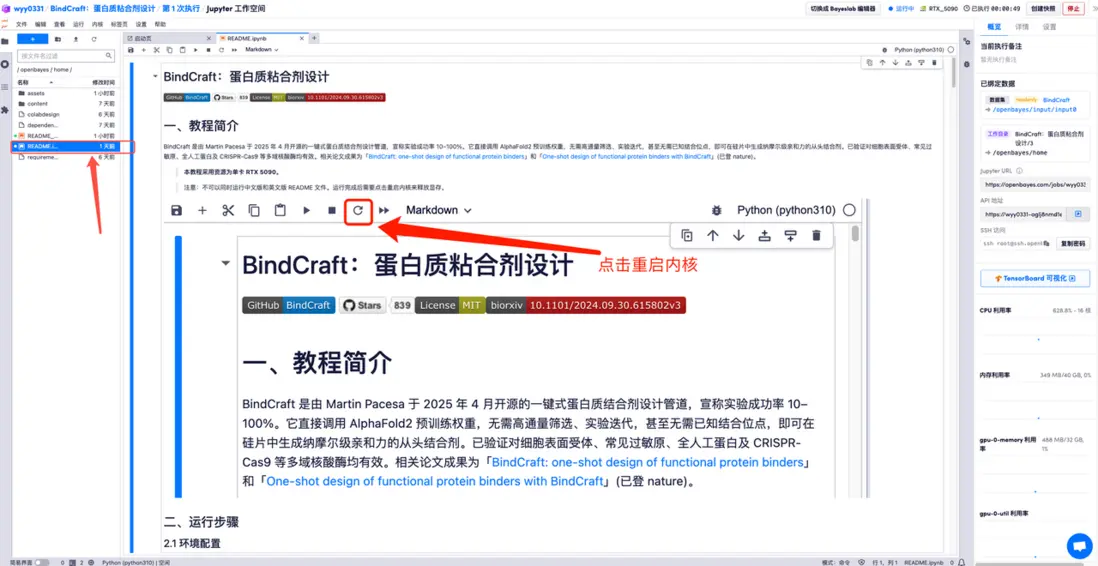
5.以下為 BindCraft 使用頁面,點擊「README」即可跳轉至生成界面。
02 效果演示
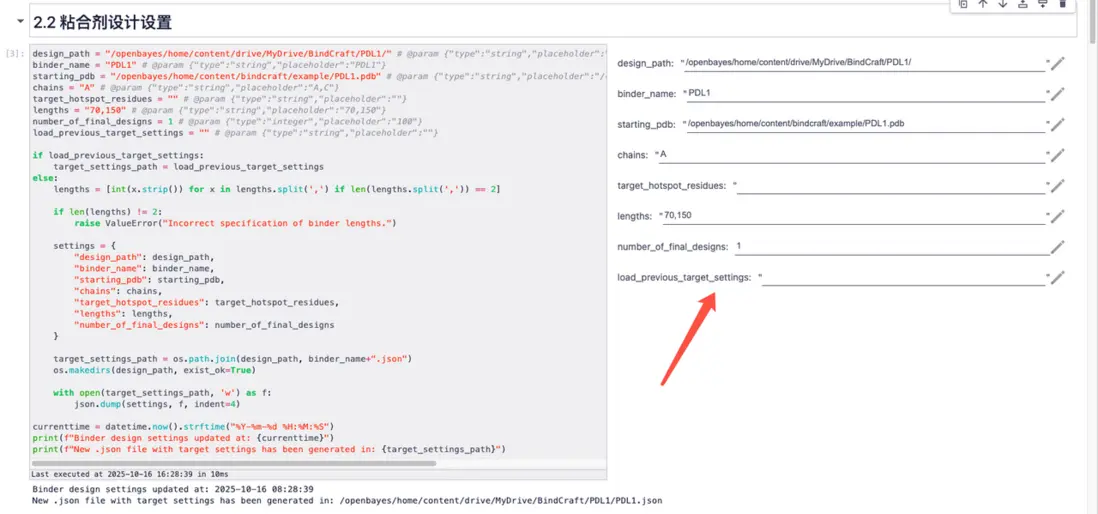
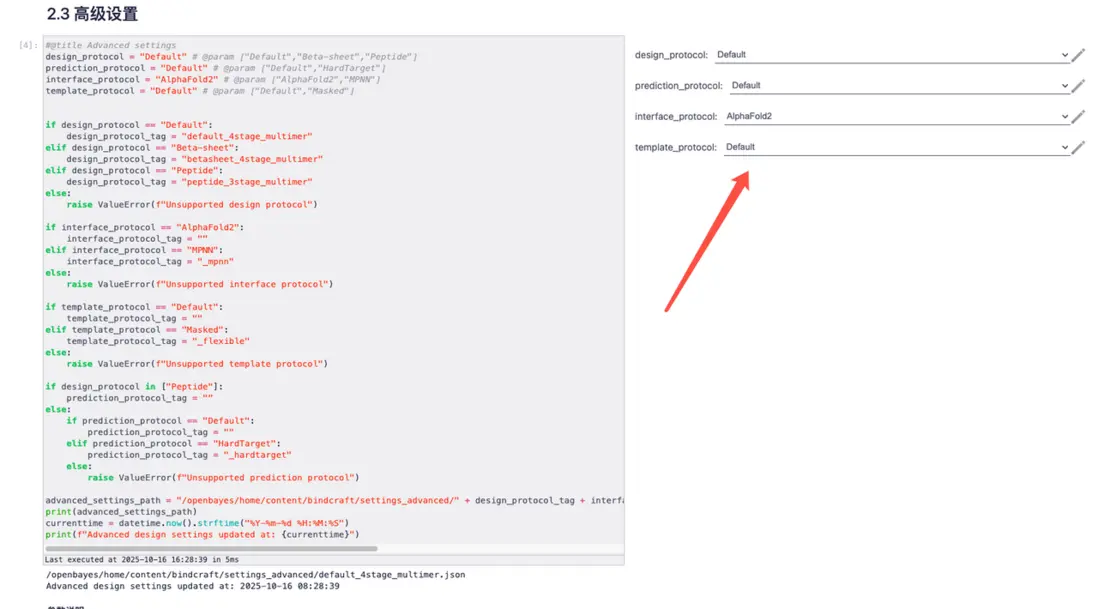
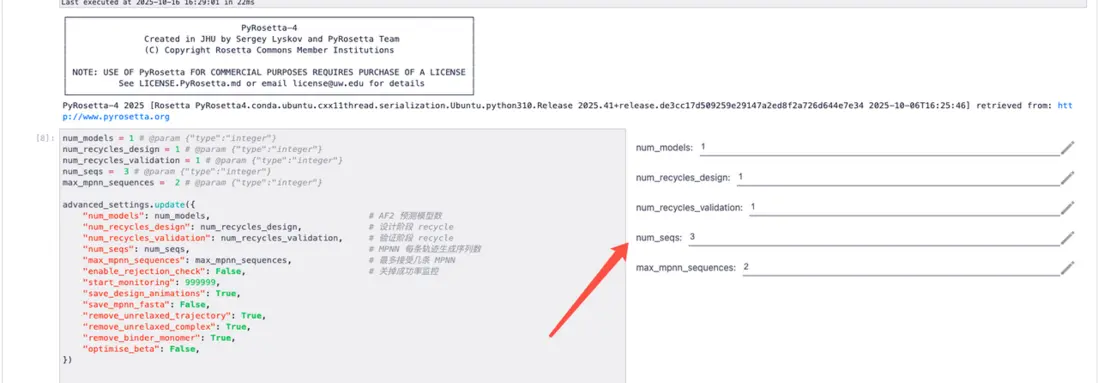
請根據 README 參數説明完成相關參數設置。
以默認參數為例,相關效果如下所示。
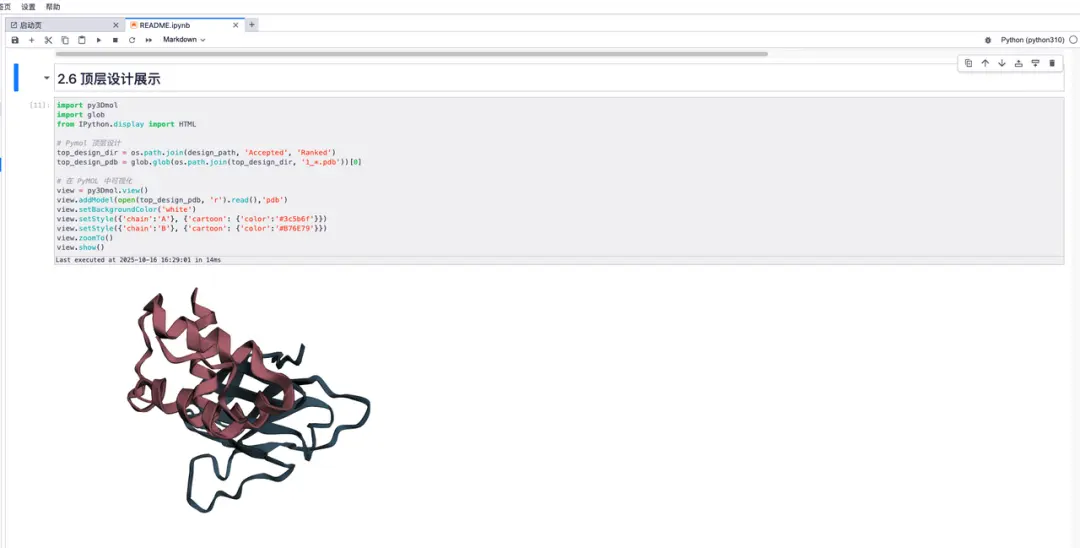
「頂層設計」效果,如下圖: