在常規的基因組學研究中,人們常常把“參照基因組”當作某一物種遺傳信息的“規範模板”。但這猶如僅通過一份標準住宅設計圖去推斷所有家庭的住宅佈局,不可避免地忽視了眾多個性化的特徵。泛基因組(Pan-genome)概念的提出,完全超越了這一限制,為我們展現了一個更加宏大、精確且不斷變化的物種遺傳全貌。
本期將聚焦泛基因組在遺傳疾病相關研究中的優勢。
一、突破“單一參考”的侷限:從個體代表性到羣體包容性
自2003年人類基因組計劃(HGP)完成以來,GRCh38等線性參考基因組雖為基因組學研究奠定了基石,但其本質是一個拼接自少數個體(且以歐洲血統為主)的“共識”序列,無法反映人類羣體內部巨大的遺傳多態性。尤其在複雜區域——如着絲粒周邊(pericentromeric)、端粒附近(subtelomeric)、節段重複序列(segmental duplications, SDs)及rDNA陣列中——仍存在約150 Mb的缺口。即便2022年T2T-CHM13實現了首個端粒到端粒(T2T)無缺口單倍體組裝,它仍是單一單倍型,無法代表結構變異(SVs)的羣體多樣性。
HPRC(人類泛基因組參考聯盟)基於47個全球分佈個體(94條單倍型)構建的泛基因組,首次實現了對人類基因組“常見變異空間”的系統性覆蓋,從根本上解決了傳統參考中“參考偏向”(reference bias)問題——即非參考等位基因在比對中被系統性低估或誤判。
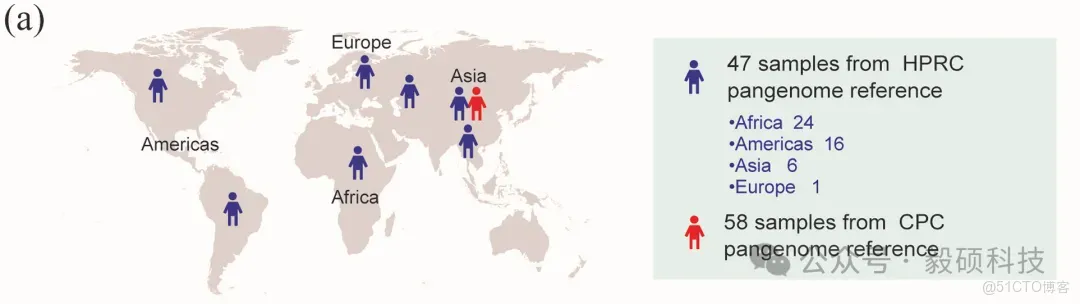
圖1 兩個泛基因組項目所採集樣本的地理來源
- HPRC(人類泛基因組參考聯盟):47個個體,來自非洲、美洲、亞洲、歐洲四大洲;
- CPC(中國泛基因組聯盟):58個核心樣本,覆蓋中國36個少數民族。
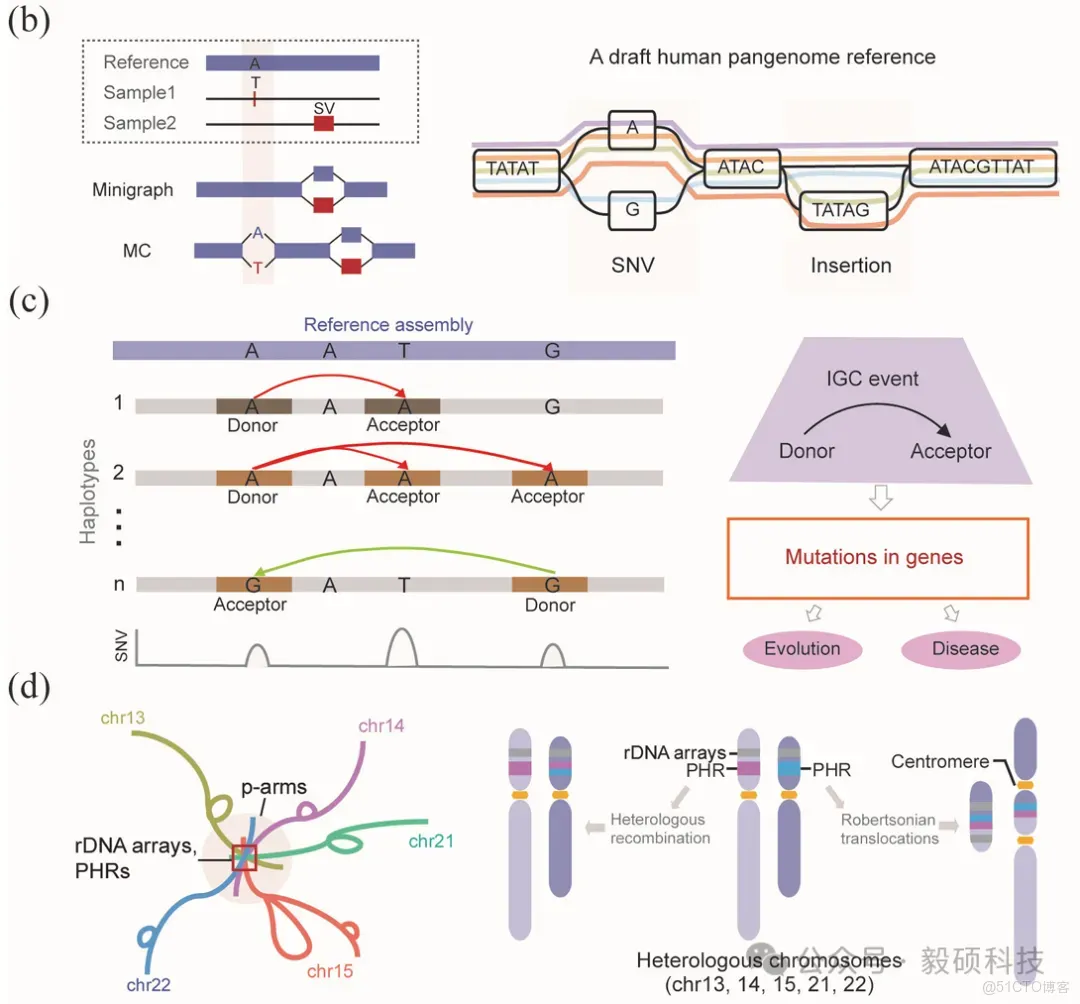
泛基因組的核心理念在於:用圖結構(graph-based pangenome)替代線性序列,整合多個高質量、單倍型分型(haplotype-phased)的個體基因組,構建一個能容納多種等位基因、插入缺失、倒位、拷貝數變異等的“參考網絡”。
泛基因組圖譜能容納SNV、Indel、CNV、倒位等各類變異,能覆蓋節段重複序列(SDs)這一傳統“盲區。從機制上解釋了新生兒中高達1/800的羅伯遜易位(Robertsonian translocations, ROBs)發生率,證明泛基因組可解析染色體結構病的起源。
相比GRCh38,新增119 Mb 常見多態序列,新增1,115 個新基因重複(novel gene duplications),其中約90 Mb 源於結構變異(SVs)。
二、顯著提升變異檢出能力與準確性
23年的一篇綜述[1]明確指出,以泛基因組替代GRCh38作為比對參考後,變異檢測性能獲得飛躍式提升:
- 小變異(SNV/Indel)檢測錯誤率降低34%:因圖結構可將 reads 更準確地錨定至其真實來源的等位路徑,避免了在高度同源區域(如SDs)的錯配。
- 每單倍型檢出的結構變異數量增加104%:即翻倍!這意味着大量以往“不可見”的SVs(尤其是複雜SVs,如嵌套插入、倒位伴隨缺失)被系統性揭示。例如,HPRC泛基因組新增了119 Mb的常見多態序列和1,115個新基因重複——其中約90 Mb源於SVs。
具體來看,泛基因組的最大價值之一,在於它首次使得對高度重複、傳統上難以比對的區域進行系統性羣體遺傳學研究成為可能:
1. 節段重複區(SDs)是SNV富集與基因轉換熱點
Vollger et al.(2023)利用HPRC泛基因組發現:SDs中的SNV密度比非重複區高1.6倍,其中至少23%歸因於基因間基因轉換(Interlocus Gene Conversion, IGC)。不同於等位基因間的同源轉換,IGC發生在不同基因座之間,可導致功能基因(如F8凝血因子、HBG1胎兒血紅蛋白、C4B補體)的等位基因“橫向傳播”,既可能促進有利突變擴散(進化意義),也可能造成致病突變蔓延(疾病風險)。研究識別出800餘個受IGC影響的蛋白編碼基因,其中38個為進化上高度保守的“約束基因”(constrained genes),突變易致嚴重表型。
2. 近端着絲粒染色體短臂(acrocentric p-arms)是異源重組温牀
Guarracino et al.(2023)通過泛基因組圖譜發現:13、14、15、21、22號染色體的p-臂因富含rDNA陣列與偽同源區(PHRs),在減數分裂中易發生非同源染色體間的異源重組(heterologous recombination),直接導致羅伯遜易位(ROBs)——新生兒中發生率約1/800。泛基因組首次精準定位ROBs斷點均位於PHRs,從機制上解釋了這類常見染色體病的起源。
這些區域曾是臨牀基因組學的“禁區”,如今泛基因組正將其轉化為新的致病機制與生物標誌物發現源泉。
三、推動精準醫學與人羣特異性研究:以中國泛基因組(CPC)為例
HPRC泛基因組雖具開創性,但亞洲樣本僅佔13%,存在顯著人羣偏差。對此,中國泛基因組聯盟(CPC)率先響應,基於58個核心樣本(涵蓋36個少數民族),構建了首箇中國人羣代表性泛基因組:
- 新增189 Mb多態序列、1,367個重複蛋白編碼基因。
- 發現78,072個SVs,其中34,223個為全新變異。
- 鑑定出與角質化、紫外線響應、DNA修復、免疫及壽命調控相關的新基因/等位基因,部分源於古菌水平基因轉移。
尤為關鍵的是,CPC數據成功將558個ClinVar中列為“致病/可能致病/VUS”的變異重分類為“可能良性”——因其在東亞人羣中頻率過高(而既往公共數據庫缺乏亞洲數據)。這直接避免了臨牀誤診,彰顯泛基因組在人羣特異性變異解讀中的不可替代性:沒有足夠多樣化的泛基因組,罕見病診斷與遺傳諮詢將長期受制於“歐洲中心主義”偏見。
參考文獻
[1] Wang, B., Dang, N., Yang, X., Xu, S., & Ye, K. (2023). The human pangenome reference: the beginning of a new era for genomics. Science Bulletin, 68(14), 1484–1487.
Sentieon軟件介紹
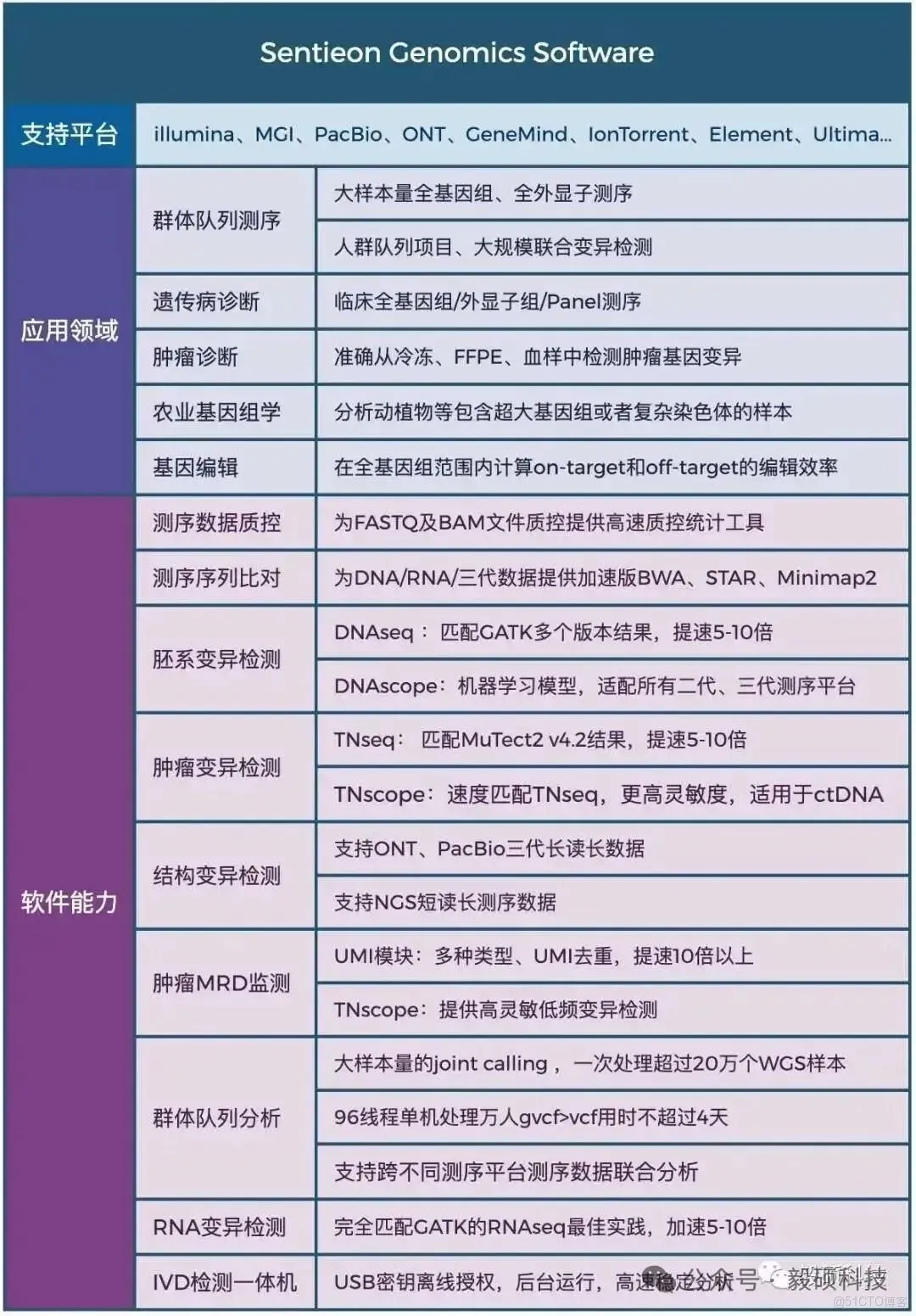
Sentieon為完整的純軟件基因變異檢測二級分析方案,其分析流程完全忠於BWA、GATK、MuTect2、STAR、Minimap2、Fgbio、picard等金標準的數學模型。在匹配開源流程分析結果的前提下,大幅提升WGS、WES、Panel、UMI、ctDNA、RNA等測序數據的分析效率和檢出精度,並匹配目前全部第二代、三代測序平台。
Sentieon軟件團隊擁有豐富的軟件開發及算法優化工程經驗,致力於解決生物數據分析中的速度與準確度瓶頸,為來自於分子診斷、藥物研發、臨牀醫療、人羣隊列、動植物等多個領域的合作伙伴提供高效精準的軟件解決方案,共同推動基因技術的發展。
截至2025年7月份,Sentieon已經在全球範圍內為1860+用户提供服務,用户處理超過4980+PB數據量,被世界一級影響因子刊物如NEJM、Cell、Nature等廣泛引用,引用次數超過1500篇。此外,Sentieon連續數年摘得了Precision FDA、Dream Challenges等多個權威評比的桂冠,在業內獲得廣泛認可。